Eine Deep-Learning-Pipeline zur Kontrolle von Proteininteraktionen
Im Jahr 2023 veröffentlichten Wissenschaftler des gemeinsamen Labors für Proteindesign und Immunoengineering (LPDI) der School of Engineering und der School of Life Sciences unter der Leitung von Bruno Correia in Nature eine Deep-Learning-Pipeline für die Entwicklung neuer Proteine, die mit therapeutischen Zielen interagieren. MaSIF ist in der Lage, Millionen von Proteinen schnell zu scannen, um optimale Übereinstimmungen zwischen Molekülen auf der Grundlage ihrer chemischen und geometrischen Oberflächeneigenschaften zu ermitteln, wodurch Wissenschaftler in die Lage versetzt werden, neuartige Protein-Protein-Wechselwirkungen zu entwickeln, die eine Schlüsselrolle bei der Zellregulierung und bei Therapeutika spielen.
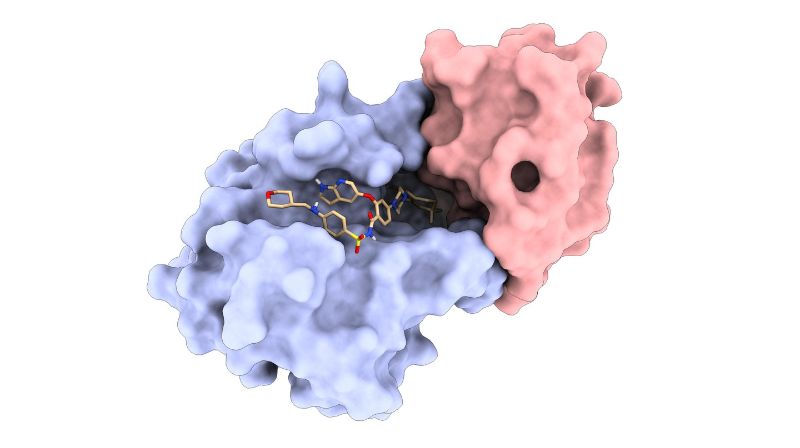
Eineinhalb Jahre später hat das Team – wiederum in Nature – über eine spannende Weiterentwicklung dieser Technologie berichtet. Sie haben MaSIF eingesetzt, um neuartige Proteinbinder zu entwickeln, die mit bekannten Proteinkomplexen interagieren, an denen kleine Moleküle wie therapeutische Medikamente oder Hormone beteiligt sind. Da diese gebundenen kleinen Moleküle subtile Veränderungen der Oberflächeneigenschaften («Neooberflächen») dieser Protein-Wirkstoff-Komplexe bewirken, können sie als «An»- oder «Aus»-Schalter für die Feinsteuerung von Zellfunktionen wie DNA-Transkription oder Proteinabbau fungieren.
«Unsere Idee war es, eine Wechselwirkung zu entwickeln, bei der ein kleines Molekül zwei Proteine zusammenbringt. Einige Ansätze haben sich auf die Suche nach solchen kleinen Molekülen konzentriert, aber wir wollten ein neuartiges Protein entwerfen, das an einen definierten Protein-Wirkstoff-Komplex bindet», sagt LPDI-Wissenschaftler und Co-Erstautor Anthony Marchand.
Bemerkenswerterweise konnte das Team zeigen, dass MaSIF die Repräsentationen von Proteinoberflächen («Fingerabdrücke»), die nur auf Proteine trainiert worden waren, nahtlos auf Neooberflächen von Protein-Wirkstoff-Komplexen anwenden konnte. Während die meisten lernbasierten Proteindesignsysteme nur mit Aminosäurebausteinen aus der Natur arbeiten, bedeutet die Empfindlichkeit und Verallgemeinerbarkeit von MaSIF für kleine Moleküle, dass es für das Design chemisch induzierter Proteininteraktionen in künstlichen Zellen für medikamentengesteuerte zellbasierte Therapien oder Biosensoren eingesetzt werden könnte.
Klein aber fein
Die Bindung von Proteinen scheint zwar so einfach zu sein wie das Zusammenfügen von Puzzleteilen, doch in Wirklichkeit ist es aufgrund von Variationen der Proteinoberfläche schwer vorherzusagen, wie und wo Bindungsvorgänge stattfinden werden. Wie in ihrer früheren Studie entwarf das Team neuartige Proteinbindemittel, indem es mit MaSIF «Fingerabdrücke» für Oberflächenmerkmale wie positive und negative Ladung, Hydrophobizität, Form usw. erstellte. Dann identifizierten sie komplementäre Oberflächen aus einer Datenbank, pfropften Proteinfragmente digital auf grössere Gerüste und wählten Bindemittel aus, von denen sie annahmen, dass sie am besten zu ihren Zielen passen.
«Der Unterschied besteht darin, dass wir davon ausgehen, dass sich die Oberflächeneigenschaften eines Proteins ändern, wenn ein kleines Molekül daran bindet, wodurch eine neue Oberfläche entsteht. MaSIF war in der Lage, diesen Unterschied mit einem hohen Maß an Empfindlichkeit zu erfassen», sagt LPDI-Doktorand und Mitautor Arne Schneuing.
Das Team validierte seine neuartigen Proteinbinder experimentell an drei medikamentengebundenen Proteinkomplexen, die das Hormon Progesteron, das von der FDA zugelassene Leukämiemedikament Venetoclax bzw. das natürlich vorkommende Antibiotikum Actinonin enthalten. Die mit Hilfe von MaSIF entwickelten Proteinbinder erkannten jeden Wirkstoff-Protein-Komplex erfolgreich und mit hoher Affinität. Die Forscher erklären, dass dies möglich war, weil MaSIF auf allgemeinen Oberflächenmerkmalen basiert, die sowohl für Proteine als auch für kleine Moleküle gelten, so dass sie die Merkmale der kleinen Moleküle auf denselben Deskriptorraum abbilden konnten, auf den MaSIF für Proteine trainiert wurde.
«MaSIF hat eine relativ kleine Anzahl von Parametern – etwa 70 000 im Vergleich zu Milliarden bei grossen Deep-Learning-Systemen wie ChatGPT. Dies ist möglich, weil wir nur wichtige Oberflächenmerkmale verwenden, was zu einem hohen Abstraktionsgrad führt. Mit anderen Worten, wir geben dem System nicht das gesamte Bild, sondern nur den Teil, den wir für die Lösung des Problems für wichtig halten», sagt Schneuing.
Bessere Kontrolle von CAR-T-Zellen
Eine spannende potenzielle Anwendung dieser Arbeit ist die Feinsteuerung zellbasierter Krebsbehandlungen wie der CAR-T-Therapie (chimäre Antigenrezeptoren), bei der die T-Zellen eines Patienten so verändert werden, dass sie den Krebs besser bekämpfen. Nach der Wiedereinführung in den Patienten können die manipulierten Zellen jedoch die falschen Ziele angreifen – was zu schädlichen Nebenwirkungen führen kann – oder ihre Fähigkeit zur Krebsbekämpfung erschöpfen. In einem Proof-of-Concept-Experiment zeigte das EPFL-Team, dass ein mit MaSIF entwickeltes System, das Venetoclax induzieren kann, die tumortötende Aktivität von CAR-T-Zellen in vitro wirksam einschalten kann.
«Wenn man die räumlich-zeitliche Aktivität von zellbasierten Therapien mit kleinen Molekülschaltern präzise steuern kann, dann kann man die Sicherheit und Wirksamkeit der Behandlung wirklich verbessern», fasst LPDI-Doktorand und Erstautor Stephen Buckley zusammen.
Kollaboration
Diese Arbeit wurde in Zusammenarbeit mit Michael Bronstein, dem wissenschaftlichen Leiter des Forschungsinstituts für Biomedizinische Künstliche Intelligenz (AITHYRA) der Österreichischen Akademie der Wissenschaften, durchgeführt.