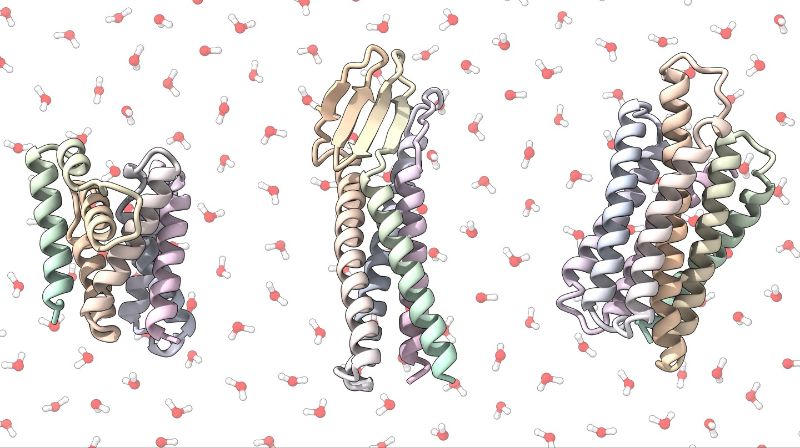
Analoga von Membranproteinen für die biomedizinische Forschung
Viele Wege zur Entdeckung von Arzneimitteln und Antikörpern konzentrieren sich auf kompliziert gefaltete Zellmembranproteine: Wenn Moleküle eines Arzneimittelkandidaten an diese Proteine binden, wie ein Schlüssel an ein Schloss, lösen sie chemische Kaskaden aus, die das Zellverhalten verändern. Da diese Proteine jedoch in die lipidhaltige äussere Schicht der Zellen eingebettet sind, sind sie nur schwer zugänglich und in wasserbasierten Lösungen unlöslich (hydrophob), was ihre Untersuchung erschwert.
«Wir wollten diese Proteine aus der Zellmembran herausholen, also haben wir sie als hyperstabile, lösliche Analoga umgestaltet, die wie Membranproteine aussehen, aber viel einfacher zu bearbeiten sind», erklärt Casper Goverde, Doktorand im Laboratory of Protein Design and Immunoengineering (LPDI) der Fakultät für Ingenieurwesen und Technologie.
Kurz gesagt, Goverde und ein Forschungsteam im LPDI unter der Leitung von Bruno Correia verwendeten Deep Learning, um synthetische lösliche Versionen von Zellmembranproteinen zu entwerfen, die häufig in der pharmazeutischen Forschung verwendet werden. Während herkömmliche Screening-Methoden auf der indirekten Beobachtung zellulärer Reaktionen auf Wirkstoff- und Antikörperkandidaten oder der mühsamen Extraktion kleiner Mengen von Membranproteinen aus Säugetierzellen beruhen, ermöglicht es der rechnergestützte Ansatz der Forschenden, Zellen aus der Gleichung zu entfernen. Nachdem sie mit Hilfe ihrer Deep-Learning-Pipeline ein lösliches Proteinanalogon entworfen haben, können sie Bakterien verwenden, um das modifizierte Protein in grossen Mengen zu produzieren. Diese Proteine können dann direkt in Lösung mit den molekularen Kandidaten von Interesse binden.
«Wir schätzen, dass die Herstellung einer Charge löslicher Proteinanaloga mit E. coli etwa zehnmal weniger kostet als mit Säugetierzellen», fügt Doktorand Nicolas Goldbach hinzu.
Das Drehbuch für das Proteindesign umkehren
In den letzten Jahren haben Wissenschaftlerinnen und Wissenschaftler erfolgreich Netzwerke künstlicher Intelligenz eingesetzt, die tiefes Lernen verwenden, um neue Proteinstrukturen zu entwerfen, indem sie diese beispielsweise auf der Grundlage einer eingegebenen Sequenz von Aminosäurebausteinen vorhersagen. Für diese Studie interessierten sich die Forschenden jedoch für Proteinfalten, die bereits in der Natur vorkommen; was sie brauchten, war eine besser zugängliche, lösliche Version dieser Proteine.
«Wir hatten die Idee, diese Deep-Learning-Pipeline, die Proteinstrukturen vorhersagt, umzukehren: Wenn wir eine Struktur eingeben, kann sie uns dann die entsprechende Aminosäuresequenz sagen?», erklärt Goverde.
Um dies zu erreichen, verwendete das Team die Strukturvorhersageplattform AlphaFold2 von Google DeepMind, um Aminosäuresequenzen für lösliche Versionen mehrerer wichtiger Zellmembranproteine auf der Grundlage ihrer 3D-Struktur zu erstellen. Dann verwendeten sie ein zweites Deep-Learning-Netzwerk, ProteinMPNN, um diese Sequenzen für funktionale, lösliche Proteine zu optimieren. Die Forschenden entdeckten, dass ihr Ansatz bemerkenswerten Erfolg und Genauigkeit bei der Herstellung löslicher Proteine zeigte, die Teile ihrer nativen Funktionalität beibehielten, selbst wenn sie auf hochkomplexe Faltungen angewendet wurden, die sich bisher anderen Designmethoden entzogen.
«Der heilige Gral der Biochemie»
Ein besonderer Erfolg der Studie war der Erfolg der Pipeline bei der Entwicklung eines löslichen Analogons einer Proteinform, die als G-Protein-gekoppelter Rezeptor (GPCR) bekannt ist, der etwa 40 % der menschlichen Zellmembranproteine ausmacht und ein wichtiges pharmazeutisches Ziel darstellt.
«Wir haben zum ersten Mal gezeigt, dass wir die GPCR-Form in ein stabiles lösliches Analogon umgestalten können. Dies ist seit langem ein Problem in der Biochemie, denn wenn man es löslich machen kann, kann man viel schneller und einfacher nach neuen Medikamenten suchen», sagt LPDI-Wissenschaftler Martin Pacesa.
Die Forschenden sehen diese Ergebnisse auch als Konzeptnachweis für die Anwendung ihrer Pipeline in der Impfstoffforschung und sogar für Krebstherapeutika. So haben sie beispielsweise ein lösliches Analogon eines Proteintyps namens Claudin entwickelt, der eine Rolle dabei spielt, Tumore resistent gegen das Immunsystem und die Chemotherapie zu machen. In ihren Experimenten behielt das lösliche Claudin-Analogon des Teams seine biologischen Eigenschaften bei, was das Versprechen der Pipeline untermauert, interessante Ziele für die pharmazeutische Entwicklung zu generieren.